Avenir de l’intelligence artificielle en anatomie pathologique
Les techniques d’IA sont en plein développement dans le domaine de la santé, notamment en oncologie. L’apprentissage en profondeur (deep learning) et les réseaux neuronaux artificiels (RNA) sont des outils informatiques permettant une gestion et une analyse automatisée et prédictive de données de plus en plus abondantes. Les multiples avancées en biologie moléculaire, en génomique et en pathologie numérique aboutissent à un nombre gigantesque d’informations pour lesquelles ces nouvelles technologies seront utiles. En effet, la numérisation des lames permet le développement d’outils basés sur l’IA et plus particulièrement des RNA dont les deux applications principales sont la classification morphologique et la régression pour le pronostic médical. Cet article explique le fonctionnement des RNA et décrit ensuite leurs applications dans le domaine de l’anatomopathologie.

Sommaire
Fonctionnement des RNA
Les RNA et leurs fonctionnements sont inspirés des réseaux de neurones du cerveau humain et suivent des architectures et des modèles mathématiques plus ou moins complexes en fonction de leurs objectifs [Figures 1 & 2].
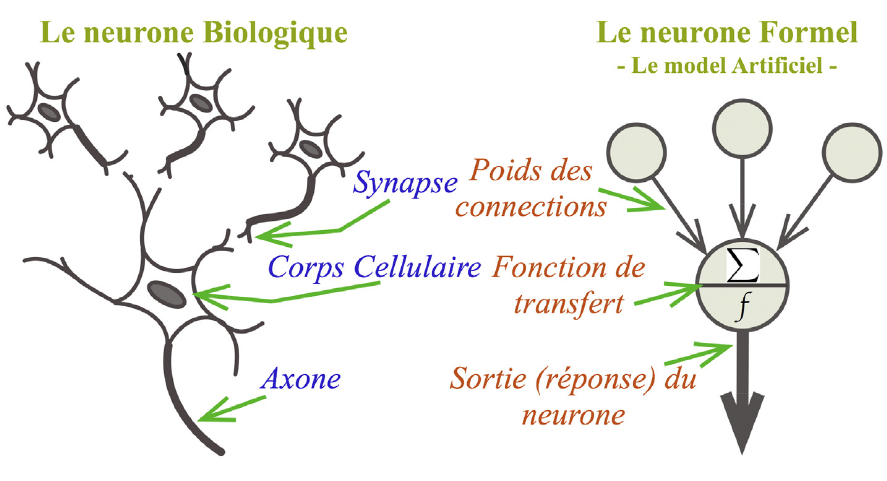
[Figure 1] Similitude entre un neurone biologique et artificiel
Un RNA est composé de neurones répartis en trois couches : une couche d’entrée, une couche cachée et une couche de sortie. Chaque neurone peut être connecté à un ou plusieurs neurones, chaque connexion en amont et en aval d’un neurone est pondérée par le poids des connexions. La transmission (forward propagation) des réponses reçues par le neurone aux neurones suivant dépend du traitement effectué par le neurone en question : l’activation ou non de sa fonction d’activation (fonction non linéaire de la somme des entrées pondérées). Plus le nombre de neurones est élevé, plus l’apprentissage de ce réseau est considéré comme profond.
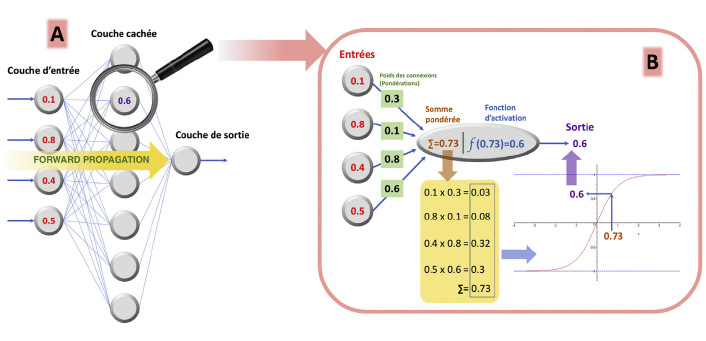
[Figure 2]
A. Représentation d’un réseau de neurones à une couche cachée
B. Représentation du traitement de l’information d’un neurone artificiel
La complexité des analyses d’image requiert des RNA avec de nombreux neurones et de nombreuses couches cachées (deep neural networks), reposant sur l’utilisation d’ordinateurs très puissants. Les RNA sont donc des outils informatiques qui fonctionnent avec une phase d’apprentissage profond (deep learning algorithm) et une phase d’exploitation.
Phases d’apprentissage et d’exploitation
La phase d’apprentissage est essentielle et nécessite une collaboration cross-expertise pour construire un modèle performant sur le plan scientifique et technique. Le RNA apprend à construire son modèle lors de cette phase en fonction de la complexité des données en entrée du réseau et de l’objectif en sortie. Cet apprentissage (supervisé ou non) consiste à minimiser les erreurs en équilibrant au mieux les poids des différentes connexions et se déroule en trois étapes. Tout d’abord, des cas d’école avec résultats sont intégrés lors de l’étape initiale d’apprentissage. Puis, de nouveaux cas sont proposés sans les résultats. Le calcul de l’erreur correspond alors à la confrontation du résultat obtenu avec celui attendu (déterminé par l’expert, le pathologiste). Ensuite, la back propagation permet le rééquilibrage des différents poids des connexions interneuronales pour minimiser l’erreur et ainsi améliorer la prédiction du modèle. L’apprentissage progresse à chaque répétition de ces cycles d’apprentissages supervisés. Dans le cadre d’une application de classification, une matrice de confusion est utilisée pour valider la phase d’apprentissage. Le degré d’exactitude de la classification de cas connus par le RNA est ainsi mesuré. Enfin, le degré de maturité du modèle est déterminé par l’expert sur des cas inconnus.
La qualité de l’apprentissage dépend donc de nombreux paramètres comme la quantité de cas disponibles pour cette phase. L’enjeu est donc de déterminer un nombre de cas suffisant pour de bonnes performances en phase d’exploitation comme outil d’aide au diagnostic par exemple [Figure 3].
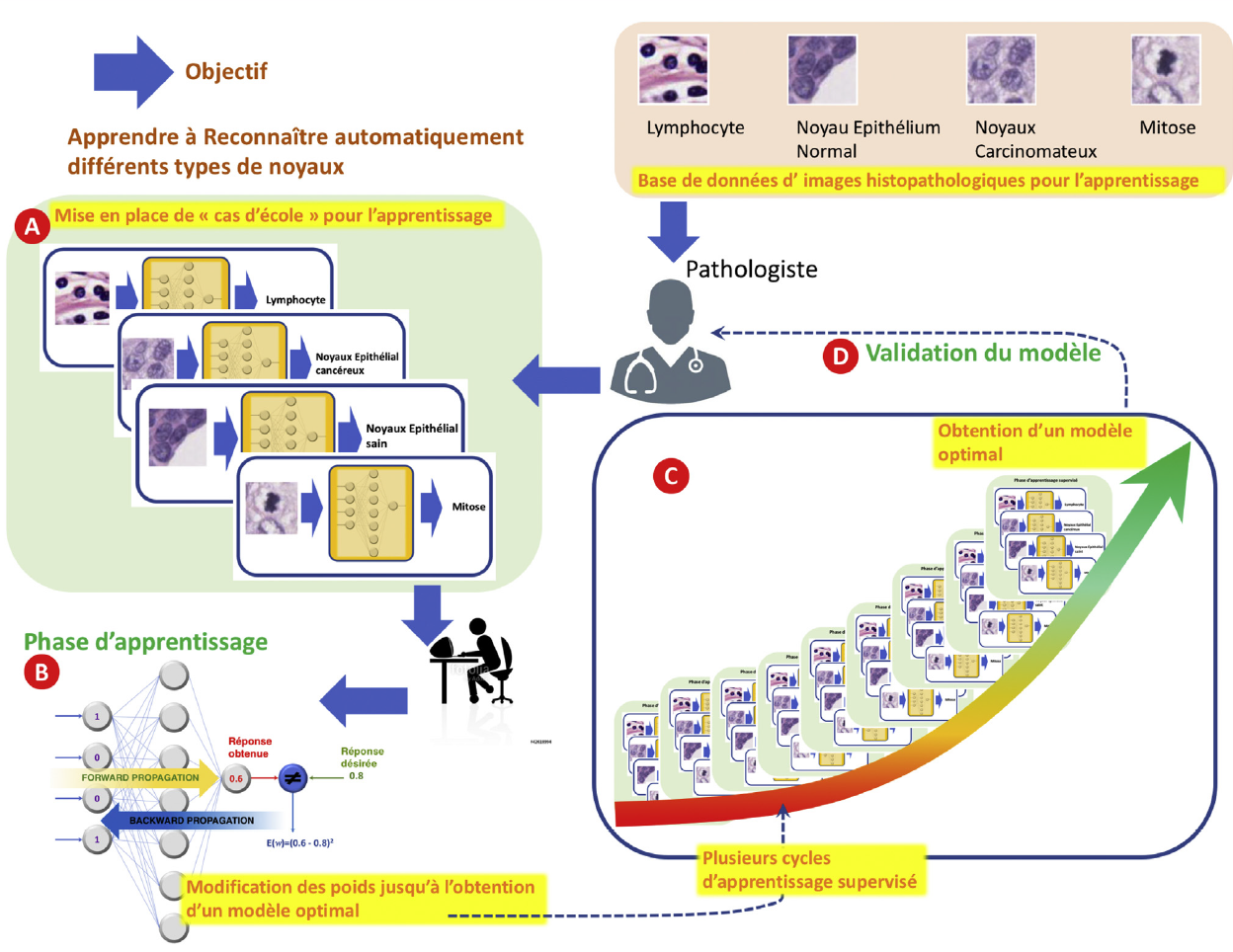
[Figure 3]
Représentation des différentes étapes de la construction d’un RNA
A. Sélection et introduction des cas d’école
B. Apprentissage supervisé, forward et backward propagation de l’information pour améliorer le modèle en ajustant le poids des connexions
C. Recommencer ces cycles d’apprentissages
D. Degré de maturation du modèle par le pathologiste.
Classification et régression
Les applications en anatomopathologie sont possibles grâce au développement de la numérisation des lames (scanner whole slide image, WSI). Les deux principales applications sont la classification et la régression.
La classification permet de catégoriser des données en fonction de classifications préétablies, par exemple une reconnaissance de formes permettant une détection, une segmentation et une classification automatique des différents types de noyaux, après un apprentissage supervisé [Figure 3]. Le principal enjeu pour les RNA de cette phase de diagnostic est l’identification des region of interest (ROI).
Quelques exemples sur WSI :
- Le faible grossissement permet la reconnaissance globale de carcinome infiltrant pour identifier automatiquement l’étendue d’une tumeur invasive 2;
- Le fort grossissement permet le dénombrement des mitoses pour la quantification de l’index mitotique3 ou encore l’analyse de l’expression d’HER2 à visée théranostique5.
La régression utilise des données plutôt quantitatives (âge du patient, taille de la tumeur, taux de prolifération…) pour établir, par exemple, un pronostic ou déterminer un score de récurrence. La détermination du score Oncotype DX dans les carcinomes mammaires à partir de critères histopronostiques (âge, taille, statut ganglionnaire, SBR, RE, RP, ki67, HER2) peut être prise pour exemple [4].
Un avenir possible de l’IA en anatomopathologie ?
Les développements de RNA, tant en termes d’outils d’analyse d’images de diagnostic assistés par ordinateur (computer aided diagnosis, CAD) ou de gestion de données de plus en plus nombreuses (Big Data), pourront, en pratique, bénéficier aux anatomopathologistes. Le développement de ces nouvelles technologies est en cours et l’implication des anatomopathologistes dans cette démarche est essentielle pour, d’une part, construire et valider les modèles de RNA et, d’autre part, accompagner et faire évoluer au cours du temps ces outils et leur application en pratique.
Glossaire
- Couche de neurones cachés : couche de neurones comprise entre la couche d’entrée et de sortie
- Back propagation : rétrodiffusion de l’information afin d’équilibrer le poids des connexions, de minimiser les erreurs et d’améliorer la prédiction
- Matrice de confusion : outil permettant d’évaluer la qualité de l’apprentissage d’un RNA pour la classification
- Poids d’une connexion : paramètre ajustable de la liaison entre deux neurones
- Region of interest : partie d’un ensemble de donnée identifiée pour un but particulier
Références
(1) Zemouri R, Devalland C, Valmary-DeganoS, Zerhouni N. Intelligence artificielle : quel avenir en anatomie pathologique ? Annales de pathologie 2019 39, 119—129
(2) Cruz-Roa A, Gilmore H, Basavanhally A, Feldman M, Gane-san S, Shih NNC, et al. Accurate and reproducible invasive breast cancer detection in whole-slide images: a deep lear-ning approach for quantifying tumor extent. Scientic Reports 2017:7
(3) Veta M, van Diest PJ, Jiwa M, Al-Janabi S, Pluim JPW. Mitosis counting in breast cancer: object-level interobserver agreement and comparison to an automatic method. PLOS ONE 2016;11:1—13
(4) Zemouri R, Omri N, Devalland C, Arnould L, Morello B,Zerhouni N, et al. Constructive deep neural network for breastcancer diagnosis. Elsevier, ScienceDirect 2018;51:98—103
(5) Vandenberghe ME, Scott MLJ, Scorer PW, Soderberg M, Bal-cerzak D, Barker C. Relevance of deep learning to facilitate the diagnosis of her2 status in breast cancer. Scientic Rep2017;7